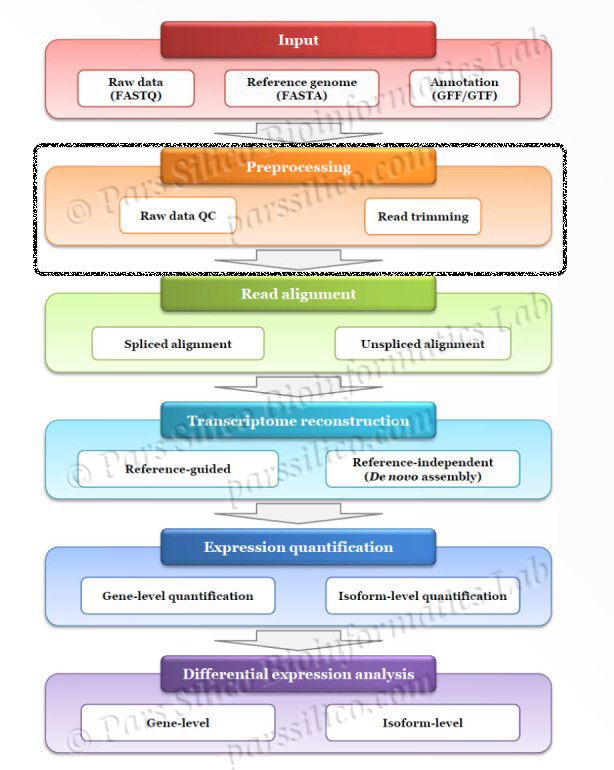
کنترل کیفیت و آماده کردن دیتاهای NGS
ارزیابی کیفیت اولین مرحله در مسیر بیوانفورماتیک RNA-seq می باشد. اغلب، برای فیلتر کردن داده، حذف توالی با کیفیت پایین و یا بازها (پیرایش)، آداپتورها ، آلودگی ها ، overrepresented و یا تصحیح اشتباهات و برای اطمینان از داشتن نتیجه نهایی دقیق ، لازم است.
معرفی ابزار های کنترل کیفیت Quality control
پکیج dupRadar : یک پکیج R برای کنترل کیفیت و انالیز میزان Duplication برای دیتا های RNA-seq
ابزار FastQC : ابزار جامعی برای کنترل کیفیت دیتا های NGS بر پایه جاوا . نسخه ویندوز لینوکس و مک موجود می باشد. فرمت فایل های ورودی میتواند BAM, SAM و FASTQ باشد
ابزار RNA-SeQC : ابزار کنترل کیفیت دیتا های RNA-seq که بر پایه جاوا می باشد. ورودی نرم فزار یک یا چند فایل BAM می باشد.
حذف اداپتور ها و فیلتر کردن
ابزار Trimmomatic : نرم افزا بر پایه جاوا که برروی هر دو نوع (single or pair-ended) فایل های fastq عمل Trimming و حذف اداپتور ها را انجام می دهد
ابزار clean_reads : ابزاری برای تمیز کردن دیتا های NGS . حذف نواحی با کیفیت پایین، اداپتور ها و..
ابزار cutadapt : ابزاری برای حذف اداپتور ها از دیتا های NGS
ابزار FASTX : بسته نرمافزاری جهت تبدیل فرمت FASTQ به FASTA ، دریافت اطلاعات کیفیت فایل و حذف و فیلتر کردن باز ها و توالی ها
پکیج ShortRead : یک پکیج R برای کنترل کیفیت دیتا ها و دستکاری و فیلتر کردن آن ها می باشد.
معرفی fastQC
توالی های پیشرفته با توان بالا می تواند ده ها میلیون توالی را در یک اجرا تولید کند. قبل از تجزیه و تحلیل این توالی برای به دست آوردن نتیجه های بیولوژیکی شما همیشه باید کنترل های کنترل کیفیت ساده را انجام دهید تا اطمینان حاصل شود که داده های خام خوب به نظر می رسند و در اطلاعات شما هیچ مشکلی وجود ندارد که ممکن است بر نحوه استفاده شما از آن تاثیر بگذارد.
اکثر ابزارهای سنج توالی، یک گزارش QC را به عنوان بخشی از pipelinتجزیه و تحلیلشان تولید می کنند، اما معمولا تنها بر شناسایی مشکلاتی که توسط خود آنها تولید ایجاد شده است، تمرکز می کنند. FastQC قصد دارد یک گزارش QC ارائه دهد که می تواند مشکلاتی را مشخص کند که در ترتیب سنج یا در مواد اولیه کتابخانه ای ایجاد می شوند.
FastQC را می توان در یکی از دو حالت زیر اجرا کرد:
- می تواند به عنوان یک برنامه تعاملی مستقل برای تجزیه و تحلیل فوری تعداد کمی از فایل های FastQ اجرا شود.
- یا می توان آن را در یک حالت غیر تعاملی اجرا کرد که در آن برای ادغام با یک خط لوله تجزیه و تحلیل بزرگتر به منظور پردازش سیستماتیک تعداد زیادی از فایل ها، مناسب است.
نصب بر روی لینوکس
FastQC یک برنامه جاوا است و برای نصب بر روی انواع سیستم عامل ها (ویندوز، لینوکس ، مک) در دسترس است. به منظور اجرای آن نیاز است که بر روی سیستم شما Java Runtime Environment (JRE) مناسب نصب شده باشد. قبل از این که سعی کنید FastQC را اجرا کنید باید اطمینان حاصل کنید که یک JRE مناسب دارید. تعدادی از JRE های مختلف در دسترس وجود دارند با این حال موارد تست شده JRE v1.6-v1.8 از اوراکل هستند. به منظور تست و آگاهی از نسخه جاوا موجود بر روی سیستم خود ، دستور زیر را در ترمینال وارد نمایید :
java –version
خروجی باید چیزی شبیه به متن زیر باشد :
java version "1.8.0_60"
Java(TM) SE Runtime Environment (build 1.8.0_60-b27)
Java HotSpot(TM) 64-Bit Server VM (build 25.60-b23, mixed mode)
در صورت عدم نصب جاوا بر روی سیستم ، به سایت java.com مراجعه کرده و نسخه مناسب، مطابق سیستم عامل خود را دانلود و نصب کنید. در اینجا میتوانید روش نصب را مشاهده کنید.
اجرای fastQC
اول از همه، شما باید به صفحه اصلی نرم افزار FastQC بروید. سپس بر روی دکمه «ownload now» کلیک کنید که باید به چند خط جلوتر بروید. در اینجا، دو راه حل وجود دارد:
1- شما می خواهید از FastQC با رابط کاربری گرافیکی استفاده کنید. برای انجام این کار، شما باید فایل FastQC را برای پلت فرم خود (ویندوز/لینوکس یا مک) انتخاب کنید. پس از آن یک installer با یک رابط کاربری گرافیکی دارید که به شما این امکان را می دهد که برنامه را به آسانی نصب کنید.
2- اگر می خواهید از FastQC با استفاده از خط فرمان استفاده کنید، بایستی گزینه Source Code for FastQC را دانلود کرده و دستورالعمل های بعدی را دنبال کنید.
نصب از کد منبع :
هنگامی که فایل را دانلود کرده و از حالت zip خارج کنید، پوشه ای با نام fastQC خواهید داشت. باید یک مکان را برای این پوشه انتخاب کنید. به عنوان مثال :
/Users/pbertin/Bioinformatics/Programs/FastQC
فایل اجرایی برنامه با نام fastqc در پوشه قرار دارد. برای اینکه بتوانید از آن استفاده کنید، باید مجوز اجرا را با استفاده از دستور زیر به آن بدهید:
chmod +x fastqc
یا
chmod 755 fastqc
با این کار شما می توانید فایل را به طور مستقیم با استفاده از دستور ./fastqc اجرا کنید.
و سپس از یک لینک نمادین در یک پوشه PATH (PATH حاوی دایرکتوری ها و فایل های اجرایی موجود در این پوشه است) استفاده نمایید برای انجام این کار میتوانید برنامه را از هر جایی از سیستم خود اجرا کنید. برای این منظور از دستور زیر استفاده کنید:
sudo ln -s /Users/pbertin/Bioinformatics/Programs/FasQC/fastqc /usr/local/bin/fastqc
(سایر ابزارهای حوزه بیوانفورماتیک را نیز همراه با توضیحاتی در مورد آنها می توانید از طریق این لینک مشاهده کنید.)
------------------------------------------------------------------
منابع : آزمایشگاه بیوانفورماتیک پارس سیلیکو ، سایت اصلی fastQC ، آموزش نصب fasteqc , سایت github ، داکیومنت راهنمای برنامه fastQC
 persian
persian English (UK)
English (UK)